Supporting information
Crystallographic Information File (CIF) https://doi.org/10.1107/S010827010500435X/dn1074sup1.cif | |
Structure factor file (CIF format) https://doi.org/10.1107/S010827010500435X/dn1074Isup2.hkl |
CCDC reference: 273028
La phase [Cu(C2O4)(C6H16N2)]·4H2O a été préparée à partir d'un mélange de nitrate de cuivre hydraté Cu(NO3)2·3H2O, du tétraméthylthylènediamine C6H16N2 et d'oxalate d'ammonium hydraté (NH4)2(C2O4)·H2O. Les réactifs pris dans les rapports molaires respectifs 1:1:2, sont placés dans un volume d'eau (15 ml). Le mélange réactionnel est mis sous agitation magnétique pendant une demi-heure. Ce mélange est enfin transvasé dans une boite de pétri recouverte. Après deux semaines d'évaporation à température ambiante, on obtient des cristaux suffisamment gros, sous forme de baguette parallélépipédique, de couleur bleue.
Les atomes d'hydrogènes des groupements CH2 et CH3 ont été introduits en position calculée et contraints de varier comme les atomes auxquels ils sont liés (C—HCH3 = 0.96 Å e t C—HCH2 = 0.97 Å). Les valeurs des facteurs de température isotropes ont été fixées par rapport aux atomes de carbone [Uiso(HCH3) = 1.5Ueq(C); Uiso(HCH2) = 1.2Ueq(C)]. Les atomes H des molécules d'eau ont été trouvés sur des séries de Fourier différence et ont été affinés avec des contraintes sur les distances O—H [0.85 (s.u.?) Å] et H···H [1.39 (s.u.?) Å] avec des facteurs isotropes Uiso(H) = 1.2Ueq(O). Le groupement diamine est partiellement desordonné autour d'un pseudo-miroir passant par l'atome N2 et le milieu de la liaison C6—C7 avec une occupation dans le rappot 0.74/0.26. Le modèle desordonné a été affiné en utilisant les outils disponibles sous SHELXL97 (Sheldrick, 1997).
Data collection: CAD-4 EXPRESS (Enraf–Nonius, 1994); cell refinement: CAD-4 EXPRESS; data reduction: MolEN (Fair, 1990); program(s) used to solve structure: SHELXS97 (Sheldrick, 1997); program(s) used to refine structure: SHELXL97 (Sheldrick, 1997); molecular graphics: ORTEP-3 for Windows (Farrugia, 1997) and PLATON (Spek, 2002); software used to prepare material for publication: SHELXL97.
| [Cu(C2O4)(C6H16N2)]·4H2O | Z = 2 |
| Mr = 339.83 | F(000) = 358 |
| Triclinic, P1 | Dx = 1.49 Mg m−3 Dm = 1.48 Mg m−3 Dm measured by flottation |
| Hall symbol: -P1 | Mo Kα radiation, λ = 0.71073 Å |
| a = 7.405 (2) Å | Cell parameters from 25 reflections |
| b = 8.890 (4) Å | θ = 10–16° |
| c = 11.883 (3) Å | µ = 1.48 mm−1 |
| α = 101.39 (3)° | T = 293 K |
| β = 98.05 (2)° | Parallelepiped, blue |
| γ = 92.70 (3)° | 0.50 × 0.21 × 0.14 mm |
| V = 757.1 (5) Å3 |
| Enraf–Nonius CAD-4 diffractometer | 2920 reflections with I > 2σ(I) |
| Radiation source: fine-focus sealed tube | Rint = 0.026 |
| Graphite monochromator | θmax = 27.0°, θmin = 1.8° |
| ω/2θ scans | h = 0→9 |
| Absorption correction: ψ scan (North et al., 1968) | k = −11→11 |
| Tmin = 0.676, Tmax = 0.794 | l = −15→15 |
| 3555 measured reflections | 2 standard reflections every 120 min |
| 3295 independent reflections | intensity decay: 1.0% |
| Refinement on F2 | Primary atom site location: structure-invariant direct methods |
| Least-squares matrix: full | Secondary atom site location: difference Fourier map |
| R[F2 > 2σ(F2)] = 0.032 | Hydrogen site location: inferred from neighbouring sites |
| wR(F2) = 0.091 | H atoms treated by a mixture of independent and constrained refinement |
| S = 1.05 | w = 1/[σ2(Fo2) + (0.0592P)2 + 0.1027P] where P = (Fo2 + 2Fc2)/3 |
| 3292 reflections | (Δ/σ)max = 0.001 |
| 240 parameters | Δρmax = 0.64 e Å−3 |
| 117 restraints | Δρmin = −0.56 e Å−3 |
| [Cu(C2O4)(C6H16N2)]·4H2O | γ = 92.70 (3)° |
| Mr = 339.83 | V = 757.1 (5) Å3 |
| Triclinic, P1 | Z = 2 |
| a = 7.405 (2) Å | Mo Kα radiation |
| b = 8.890 (4) Å | µ = 1.48 mm−1 |
| c = 11.883 (3) Å | T = 293 K |
| α = 101.39 (3)° | 0.50 × 0.21 × 0.14 mm |
| β = 98.05 (2)° |
| Enraf–Nonius CAD-4 diffractometer | 2920 reflections with I > 2σ(I) |
| Absorption correction: ψ scan (North et al., 1968) | Rint = 0.026 |
| Tmin = 0.676, Tmax = 0.794 | 2 standard reflections every 120 min |
| 3555 measured reflections | intensity decay: 1.0% |
| 3295 independent reflections |
| R[F2 > 2σ(F2)] = 0.032 | 117 restraints |
| wR(F2) = 0.091 | H atoms treated by a mixture of independent and constrained refinement |
| S = 1.05 | Δρmax = 0.64 e Å−3 |
| 3292 reflections | Δρmin = −0.56 e Å−3 |
| 240 parameters |
Geometry. All e.s.d.'s (except the e.s.d. in the dihedral angle between two l.s. planes) are estimated using the full covariance matrix. The cell e.s.d.'s are taken into account individually in the estimation of e.s.d.'s in distances, angles and torsion angles; correlations between e.s.d.'s in cell parameters are only used when they are defined by crystal symmetry. An approximate (isotropic) treatment of cell e.s.d.'s is used for estimating e.s.d.'s involving l.s. planes. |
Refinement. Refinement of F2 against ALL reflections. The weighted R-factor wR and goodness of fit S are based on F2, conventional R-factors R are based on F, with F set to zero for negative F2. The threshold expression of F2 > σ(F2) is used only for calculating R-factors(gt) etc. and is not relevant to the choice of reflections for refinement. R-factors based on F2 are statistically about twice as large as those based on F, and R- factors based on ALL data will be even larger. |
| x | y | z | Uiso*/Ueq | Occ. (<1) | |
| Cu1 | 0.14160 (3) | 0.29274 (2) | 0.134160 (18) | 0.02348 (10) | |
| C1 | 0.0474 (3) | 0.4518 (2) | −0.04893 (16) | 0.0265 (4) | |
| C2 | −0.0946 (3) | 0.0335 (2) | 0.00710 (17) | 0.0266 (4) | |
| O1 | −0.0222 (2) | 0.51464 (17) | 0.14656 (12) | 0.0318 (3) | |
| O2 | 0.1436 (2) | 0.34702 (17) | −0.02179 (12) | 0.0328 (3) | |
| O3 | −0.0943 (2) | 0.16393 (17) | 0.07127 (14) | 0.0339 (3) | |
| O4 | 0.2341 (2) | 0.04907 (17) | 0.04681 (14) | 0.0345 (3) | |
| OW1 | 0.5447 (3) | −0.0784 (3) | 0.14492 (17) | 0.0584 (5) | |
| H1W1 | 0.446 (3) | −0.045 (4) | 0.114 (3) | 0.070* | |
| H2W1 | 0.618 (4) | −0.093 (4) | 0.103 (3) | 0.070* | |
| OW2 | 0.3569 (3) | 0.0969 (2) | 0.62837 (16) | 0.0542 (5) | |
| H1W2 | 0.380 (5) | 0.098 (4) | 0.6987 (14) | 0.065* | |
| H2W2 | 0.348 (5) | 0.012 (2) | 0.587 (2) | 0.065* | |
| OW3 | 0.3272 (3) | 0.8131 (3) | 0.47040 (18) | 0.0577 (5) | |
| H1W3 | 0.221 (2) | 0.804 (4) | 0.438 (3) | 0.069* | |
| H2W3 | 0.403 (3) | 0.839 (4) | 0.432 (3) | 0.069* | |
| OW4 | −0.0356 (3) | 0.7358 (3) | 0.35038 (18) | 0.0634 (6) | |
| H1W4 | −0.042 (5) | 0.681 (3) | 0.2831 (19) | 0.076* | |
| H2W4 | −0.123 (4) | 0.790 (4) | 0.356 (3) | 0.076* | |
| N2 | 0.1339 (3) | 0.2580 (2) | 0.30008 (16) | 0.0347 (4) | |
| C7 | 0.2822 (5) | 0.3711 (5) | 0.3748 (3) | 0.0421 (8) | 0.74 |
| H71 | 0.3108 | 0.3459 | 0.4507 | 0.051* | 0.74 |
| H72 | 0.2422 | 0.4746 | 0.3848 | 0.051* | 0.74 |
| C5 | −0.0431 (7) | 0.2994 (7) | 0.3386 (7) | 0.0459 (13) | 0.74 |
| H51 | −0.1403 | 0.2299 | 0.2916 | 0.069* | 0.74 |
| H52 | −0.0642 | 0.4028 | 0.3305 | 0.069* | 0.74 |
| H53 | −0.0389 | 0.2924 | 0.4185 | 0.069* | 0.74 |
| C8 | 0.1632 (9) | 0.0998 (6) | 0.3137 (6) | 0.0526 (14) | 0.74 |
| H81 | 0.2824 | 0.0747 | 0.2967 | 0.079* | 0.74 |
| H82 | 0.0719 | 0.0298 | 0.2613 | 0.079* | 0.74 |
| H83 | 0.1543 | 0.0916 | 0.3922 | 0.079* | 0.74 |
| C6 | 0.4491 (8) | 0.3612 (7) | 0.3142 (4) | 0.0449 (14) | 0.74 |
| H61 | 0.5483 | 0.4321 | 0.3600 | 0.054* | 0.74 |
| H62 | 0.4895 | 0.2578 | 0.3045 | 0.054* | 0.74 |
| N1 | 0.3975 (3) | 0.4020 (2) | 0.19789 (16) | 0.0314 (4) | |
| C7B | 0.3293 (15) | 0.2804 (19) | 0.3551 (11) | 0.064 (4) | 0.26 |
| H7B1 | 0.3360 | 0.2951 | 0.4387 | 0.077* | 0.26 |
| H7B2 | 0.3902 | 0.1886 | 0.3286 | 0.077* | 0.26 |
| C5B | 0.008 (3) | 0.349 (2) | 0.3595 (19) | 0.068 (7) | 0.26 |
| H5B1 | −0.1145 | 0.3192 | 0.3201 | 0.102* | 0.26 |
| H5B2 | 0.0367 | 0.4562 | 0.3605 | 0.102* | 0.26 |
| H5B3 | 0.0171 | 0.3343 | 0.4378 | 0.102* | 0.26 |
| C8B | 0.084 (3) | 0.0884 (16) | 0.2951 (18) | 0.064 (6) | 0.26 |
| H8B1 | 0.1696 | 0.0273 | 0.2563 | 0.097* | 0.26 |
| H8B2 | −0.0370 | 0.0601 | 0.2533 | 0.097* | 0.26 |
| H8B3 | 0.0890 | 0.0709 | 0.3726 | 0.097* | 0.26 |
| C6B | 0.425 (3) | 0.4143 (16) | 0.3267 (12) | 0.049 (5) | 0.26 |
| H6B1 | 0.5548 | 0.4181 | 0.3557 | 0.059* | 0.26 |
| H6B2 | 0.3782 | 0.5084 | 0.3639 | 0.059* | 0.26 |
| C3 | 0.4028 (4) | 0.5703 (3) | 0.2000 (3) | 0.0535 (7) | |
| H3A | 0.3194 | 0.6168 | 0.2491 | 0.080* | |
| H3B | 0.3680 | 0.5866 | 0.1227 | 0.080* | |
| H3C | 0.5245 | 0.6161 | 0.2298 | 0.080* | |
| C4 | 0.5345 (3) | 0.3372 (3) | 0.1251 (2) | 0.0484 (6) | |
| H4A | 0.6513 | 0.3932 | 0.1537 | 0.073* | |
| H4B | 0.4965 | 0.3456 | 0.0462 | 0.073* | |
| H4C | 0.5443 | 0.2308 | 0.1286 | 0.073* |
| U11 | U22 | U33 | U12 | U13 | U23 | |
| Cu1 | 0.02363 (15) | 0.02274 (14) | 0.02366 (14) | −0.00211 (9) | 0.00184 (9) | 0.00591 (9) |
| C1 | 0.0271 (10) | 0.0272 (9) | 0.0243 (9) | −0.0025 (8) | 0.0024 (8) | 0.0048 (7) |
| C2 | 0.0236 (10) | 0.0242 (9) | 0.0312 (10) | −0.0015 (8) | 0.0022 (8) | 0.0063 (7) |
| O1 | 0.0406 (9) | 0.0339 (7) | 0.0228 (6) | 0.0050 (6) | 0.0062 (6) | 0.0089 (6) |
| O2 | 0.0394 (9) | 0.0340 (7) | 0.0273 (7) | 0.0096 (6) | 0.0071 (6) | 0.0089 (6) |
| O3 | 0.0234 (8) | 0.0292 (7) | 0.0446 (9) | 0.0000 (6) | 0.0039 (6) | −0.0018 (6) |
| O4 | 0.0218 (7) | 0.0303 (7) | 0.0468 (9) | −0.0024 (6) | 0.0007 (6) | 0.0009 (6) |
| OW1 | 0.0489 (12) | 0.0873 (16) | 0.0452 (11) | 0.0156 (11) | 0.0112 (9) | 0.0230 (10) |
| OW2 | 0.0644 (13) | 0.0545 (11) | 0.0386 (10) | 0.0025 (10) | 0.0047 (9) | −0.0003 (8) |
| OW3 | 0.0570 (13) | 0.0592 (12) | 0.0549 (12) | −0.0005 (11) | 0.0085 (10) | 0.0083 (10) |
| OW4 | 0.0682 (15) | 0.0662 (14) | 0.0470 (11) | 0.0179 (11) | 0.0017 (10) | −0.0074 (10) |
| N2 | 0.0354 (10) | 0.0422 (10) | 0.0302 (9) | 0.0008 (8) | 0.0055 (8) | 0.0162 (8) |
| C7 | 0.044 (2) | 0.052 (2) | 0.0262 (15) | −0.0024 (17) | 0.0012 (13) | 0.0041 (15) |
| C5 | 0.046 (2) | 0.056 (3) | 0.044 (3) | 0.009 (2) | 0.022 (2) | 0.020 (2) |
| C8 | 0.073 (4) | 0.048 (2) | 0.045 (3) | 0.020 (2) | 0.008 (3) | 0.0260 (19) |
| C6 | 0.034 (2) | 0.064 (4) | 0.032 (2) | −0.007 (3) | −0.0071 (15) | 0.009 (2) |
| N1 | 0.0276 (9) | 0.0322 (9) | 0.0313 (9) | −0.0060 (7) | 0.0024 (7) | 0.0022 (7) |
| C7B | 0.052 (8) | 0.100 (11) | 0.041 (6) | −0.012 (7) | −0.017 (5) | 0.039 (7) |
| C5B | 0.113 (18) | 0.071 (12) | 0.032 (8) | 0.040 (12) | 0.031 (11) | 0.020 (8) |
| C8B | 0.089 (15) | 0.072 (9) | 0.044 (8) | 0.014 (9) | 0.001 (10) | 0.046 (7) |
| C6B | 0.050 (9) | 0.043 (8) | 0.045 (7) | −0.007 (7) | −0.013 (6) | 0.004 (6) |
| C3 | 0.0441 (15) | 0.0301 (12) | 0.079 (2) | −0.0120 (11) | 0.0052 (14) | −0.0009 (12) |
| C4 | 0.0296 (13) | 0.0488 (14) | 0.0626 (16) | −0.0045 (10) | 0.0125 (11) | −0.0007 (12) |
| Cu1—O2 | 2.0069 (15) | C5—H51 | 0.9600 |
| Cu1—O3 | 2.0073 (16) | C5—H52 | 0.9600 |
| Cu1—N1 | 2.0596 (19) | C5—H53 | 0.9600 |
| Cu1—N2 | 2.0630 (18) | C8—H81 | 0.9600 |
| Cu1—O1 | 2.3549 (18) | C8—H82 | 0.9600 |
| Cu1—O4 | 2.3854 (18) | C8—H83 | 0.9600 |
| C1—O1i | 1.245 (2) | C6—N1 | 1.500 (5) |
| C1—O2 | 1.264 (3) | C6—H61 | 0.9700 |
| C1—C1i | 1.572 (4) | C6—H62 | 0.9700 |
| C2—O4ii | 1.255 (3) | N1—C4 | 1.487 (3) |
| C2—O3 | 1.255 (2) | N1—C3 | 1.490 (3) |
| C2—C2ii | 1.566 (4) | N1—C6B | 1.497 (14) |
| OW1—H1W1 | 0.864 (17) | C7B—C6B | 1.476 (14) |
| OW1—H2W1 | 0.79 (3) | C7B—H7B1 | 0.9700 |
| OW2—H1W2 | 0.826 (17) | C7B—H7B2 | 0.9700 |
| OW2—H2W2 | 0.811 (17) | C5B—H5B1 | 0.9600 |
| OW3—H1W3 | 0.821 (17) | C5B—H5B2 | 0.9600 |
| OW3—H2W3 | 0.820 (17) | C5B—H5B3 | 0.9600 |
| OW4—H1W4 | 0.845 (17) | C8B—H8B1 | 0.9600 |
| OW4—H2W4 | 0.826 (17) | C8B—H8B2 | 0.9600 |
| N2—C5B | 1.436 (13) | C8B—H8B3 | 0.9600 |
| N2—C8 | 1.470 (5) | C6B—H6B1 | 0.9700 |
| N2—C5 | 1.486 (5) | C6B—H6B2 | 0.9700 |
| N2—C7B | 1.489 (10) | C3—H3A | 0.9600 |
| N2—C7 | 1.508 (4) | C3—H3B | 0.9600 |
| N2—C8B | 1.523 (13) | C3—H3C | 0.9600 |
| C7—C6 | 1.513 (6) | C4—H4A | 0.9600 |
| C7—H71 | 0.9700 | C4—H4B | 0.9600 |
| C7—H72 | 0.9700 | C4—H4C | 0.9600 |
| O2—Cu1—O3 | 89.33 (7) | N2—C8—H83 | 109.5 |
| O2—Cu1—N1 | 92.45 (7) | N1—C6—C7 | 107.9 (4) |
| O3—Cu1—N1 | 173.55 (6) | N1—C6—H61 | 110.1 |
| O2—Cu1—N2 | 174.60 (7) | C7—C6—H61 | 110.1 |
| O3—Cu1—N2 | 92.38 (8) | N1—C6—H62 | 110.1 |
| N1—Cu1—N2 | 86.41 (8) | C7—C6—H62 | 110.1 |
| O2—Cu1—O1 | 76.80 (6) | H61—C6—H62 | 108.4 |
| O3—Cu1—O1 | 89.03 (7) | C4—N1—C3 | 107.2 (2) |
| N1—Cu1—O1 | 97.41 (7) | C4—N1—C6B | 121.9 (8) |
| N2—Cu1—O1 | 98.11 (7) | C3—N1—C6B | 96.8 (6) |
| O2—Cu1—O4 | 85.68 (7) | C4—N1—C6 | 106.3 (3) |
| O3—Cu1—O4 | 75.71 (6) | C3—N1—C6 | 114.7 (3) |
| N1—Cu1—O4 | 98.23 (7) | C4—N1—Cu1 | 110.79 (14) |
| N2—Cu1—O4 | 99.70 (8) | C3—N1—Cu1 | 112.45 (16) |
| O1—Cu1—O4 | 156.92 (6) | C6B—N1—Cu1 | 106.9 (7) |
| O1i—C1—O2 | 125.39 (19) | C6—N1—Cu1 | 105.3 (3) |
| O1i—C1—C1i | 117.4 (2) | C6B—C7B—N2 | 111.9 (11) |
| O2—C1—C1i | 117.2 (2) | C6B—C7B—H7B1 | 109.2 |
| O4ii—C2—O3 | 125.66 (19) | N2—C7B—H7B1 | 109.2 |
| O4ii—C2—C2ii | 116.6 (2) | C6B—C7B—H7B2 | 109.2 |
| O3—C2—C2ii | 117.7 (2) | N2—C7B—H7B2 | 109.2 |
| C1i—O1—Cu1 | 108.45 (13) | H7B1—C7B—H7B2 | 107.9 |
| C1—O2—Cu1 | 119.26 (13) | N2—C5B—H5B1 | 109.5 |
| C2—O3—Cu1 | 120.42 (13) | N2—C5B—H5B2 | 109.5 |
| C2ii—O4—Cu1 | 108.57 (13) | H5B1—C5B—H5B2 | 109.5 |
| H1W1—OW1—H2W1 | 113 (3) | N2—C5B—H5B3 | 109.5 |
| H1W2—OW2—H2W2 | 114 (3) | H5B1—C5B—H5B3 | 109.5 |
| H1W3—OW3—H2W3 | 115 (3) | H5B2—C5B—H5B3 | 109.5 |
| H1W4—OW4—H2W4 | 112 (2) | N2—C8B—H8B1 | 109.5 |
| C8—N2—C5 | 108.3 (4) | N2—C8B—H8B2 | 109.5 |
| C5B—N2—C7B | 116.8 (10) | H8B1—C8B—H8B2 | 109.5 |
| C8—N2—C7 | 111.3 (3) | N2—C8B—H8B3 | 109.5 |
| C5—N2—C7 | 107.7 (3) | H8B1—C8B—H8B3 | 109.5 |
| C5B—N2—C8B | 109.5 (12) | H8B2—C8B—H8B3 | 109.5 |
| C7B—N2—C8B | 103.3 (9) | C7B—C6B—N1 | 109.8 (10) |
| C5B—N2—Cu1 | 113.3 (10) | C7B—C6B—H6B1 | 109.7 |
| C8—N2—Cu1 | 114.1 (3) | N1—C6B—H6B1 | 109.7 |
| C5—N2—Cu1 | 110.9 (4) | C7B—C6B—H6B2 | 109.7 |
| C7B—N2—Cu1 | 103.8 (5) | N1—C6B—H6B2 | 109.7 |
| C7—N2—Cu1 | 104.18 (17) | H6B1—C6B—H6B2 | 108.2 |
| C8B—N2—Cu1 | 109.4 (8) | N1—C3—H3A | 109.5 |
| N2—C7—C6 | 107.6 (3) | N1—C3—H3B | 109.5 |
| N2—C7—H71 | 110.2 | H3A—C3—H3B | 109.5 |
| C6—C7—H71 | 110.2 | N1—C3—H3C | 109.5 |
| N2—C7—H72 | 110.2 | H3A—C3—H3C | 109.5 |
| C6—C7—H72 | 110.2 | H3B—C3—H3C | 109.5 |
| H71—C7—H72 | 108.5 | N1—C4—H4A | 109.5 |
| N2—C5—H51 | 109.5 | N1—C4—H4B | 109.5 |
| N2—C5—H52 | 109.5 | H4A—C4—H4B | 109.5 |
| N2—C5—H53 | 109.5 | N1—C4—H4C | 109.5 |
| N2—C8—H81 | 109.5 | H4A—C4—H4C | 109.5 |
| N2—C8—H82 | 109.5 | H4B—C4—H4C | 109.5 |
| Symmetry codes: (i) −x, −y+1, −z; (ii) −x, −y, −z. |
| D—H···A | D—H | H···A | D···A | D—H···A |
| OW1—H1W1···O4 | 0.86 (2) | 1.96 (2) | 2.820 (3) | 175 (4) |
| OW1—H2W1···O4iii | 0.79 (3) | 2.30 (3) | 3.030 (3) | 155 (3) |
| OW2—H1W2···OW1iv | 0.83 (2) | 1.91 (2) | 2.730 (3) | 172 (3) |
| OW2—H2W2···OW3v | 0.81 (2) | 2.00 (2) | 2.811 (3) | 173 (3) |
| OW3—H1W3···OW4 | 0.82 (2) | 2.03 (2) | 2.844 (3) | 169 (4) |
| OW3—H2W3···OW2vi | 0.82 (2) | 2.10 (2) | 2.907 (3) | 166 (3) |
| OW4—H1W4···O1 | 0.85 (2) | 1.99 (2) | 2.819 (3) | 165 (3) |
| OW4—H2W4···OW2vii | 0.83 (2) | 2.06 (2) | 2.879 (3) | 174 (4) |
| Symmetry codes: (iii) −x+1, −y, −z; (iv) −x+1, −y, −z+1; (v) x, y−1, z; (vi) −x+1, −y+1, −z+1; (vii) −x, −y+1, −z+1. |
Experimental details
| Crystal data | |
| Chemical formula | [Cu(C2O4)(C6H16N2)]·4H2O |
| Mr | 339.83 |
| Crystal system, space group | Triclinic, P1 |
| Temperature (K) | 293 |
| a, b, c (Å) | 7.405 (2), 8.890 (4), 11.883 (3) |
| α, β, γ (°) | 101.39 (3), 98.05 (2), 92.70 (3) |
| V (Å3) | 757.1 (5) |
| Z | 2 |
| Radiation type | Mo Kα |
| µ (mm−1) | 1.48 |
| Crystal size (mm) | 0.50 × 0.21 × 0.14 |
| Data collection | |
| Diffractometer | Enraf–Nonius CAD-4 diffractometer |
| Absorption correction | ψ scan (North et al., 1968) |
| Tmin, Tmax | 0.676, 0.794 |
| No. of measured, independent and observed [I > 2σ(I)] reflections | 3555, 3295, 2920 |
| Rint | 0.026 |
| (sin θ/λ)max (Å−1) | 0.638 |
| Refinement | |
| R[F2 > 2σ(F2)], wR(F2), S | 0.032, 0.091, 1.05 |
| No. of reflections | 3292 |
| No. of parameters | 240 |
| No. of restraints | 117 |
| H-atom treatment | H atoms treated by a mixture of independent and constrained refinement |
| Δρmax, Δρmin (e Å−3) | 0.64, −0.56 |
Computer programs: CAD-4 EXPRESS (Enraf–Nonius, 1994), CAD-4 EXPRESS, MolEN (Fair, 1990), SHELXS97 (Sheldrick, 1997), SHELXL97 (Sheldrick, 1997), ORTEP-3 for Windows (Farrugia, 1997) and PLATON (Spek, 2002), SHELXL97.
| Cu1—O2 | 2.0069 (15) | N2—C5B | 1.436 (13) |
| Cu1—O3 | 2.0073 (16) | N2—C8 | 1.470 (5) |
| Cu1—N1 | 2.0596 (19) | N2—C5 | 1.486 (5) |
| Cu1—N2 | 2.0630 (18) | N2—C7B | 1.489 (10) |
| Cu1—O1 | 2.3549 (18) | N2—C7 | 1.508 (4) |
| Cu1—O4 | 2.3854 (18) | N2—C8B | 1.523 (13) |
| C1—O1i | 1.245 (2) | C7—C6 | 1.513 (6) |
| C1—O2 | 1.264 (3) | C6—N1 | 1.500 (5) |
| C1—C1i | 1.572 (4) | N1—C4 | 1.487 (3) |
| C2—O4ii | 1.255 (3) | N1—C3 | 1.490 (3) |
| C2—O3 | 1.255 (2) | N1—C6B | 1.497 (14) |
| C2—C2ii | 1.566 (4) | C7B—C6B | 1.476 (14) |
| O2—Cu1—O3 | 89.33 (7) | O3—Cu1—O4 | 75.71 (6) |
| O2—Cu1—N1 | 92.45 (7) | N1—Cu1—O4 | 98.23 (7) |
| O3—Cu1—N1 | 173.55 (6) | N2—Cu1—O4 | 99.70 (8) |
| O2—Cu1—N2 | 174.60 (7) | O1—Cu1—O4 | 156.92 (6) |
| O3—Cu1—N2 | 92.38 (8) | O1i—C1—O2 | 125.39 (19) |
| N1—Cu1—N2 | 86.41 (8) | O1i—C1—C1i | 117.4 (2) |
| O2—Cu1—O1 | 76.80 (6) | O2—C1—C1i | 117.2 (2) |
| O3—Cu1—O1 | 89.03 (7) | O4ii—C2—O3 | 125.66 (19) |
| N1—Cu1—O1 | 97.41 (7) | O4ii—C2—C2ii | 116.6 (2) |
| N2—Cu1—O1 | 98.11 (7) | O3—C2—C2ii | 117.7 (2) |
| O2—Cu1—O4 | 85.68 (7) |
| Symmetry codes: (i) −x, −y+1, −z; (ii) −x, −y, −z. |
| D—H···A | D—H | H···A | D···A | D—H···A |
| OW1—H1W1···O4 | 0.864 (17) | 1.957 (18) | 2.820 (3) | 175 (4) |
| OW1—H2W1···O4iii | 0.79 (3) | 2.30 (3) | 3.030 (3) | 155 (3) |
| OW2—H1W2···OW1iv | 0.826 (17) | 1.909 (18) | 2.730 (3) | 172 (3) |
| OW2—H2W2···OW3v | 0.811 (17) | 2.004 (17) | 2.811 (3) | 173 (3) |
| OW3—H1W3···OW4 | 0.821 (17) | 2.034 (18) | 2.844 (3) | 169 (4) |
| OW3—H2W3···OW2vi | 0.820 (17) | 2.104 (17) | 2.907 (3) | 166 (3) |
| OW4—H1W4···O1 | 0.845 (17) | 1.994 (18) | 2.819 (3) | 165 (3) |
| OW4—H2W4···OW2vii | 0.826 (17) | 2.056 (18) | 2.879 (3) | 174 (4) |
| Symmetry codes: (iii) −x+1, −y, −z; (iv) −x+1, −y, −z+1; (v) x, y−1, z; (vi) −x+1, −y+1, −z+1; (vii) −x, −y+1, −z+1. |

 journal menu
journal menu



![[Figure 1]](http://journals.iucr.org/c/issues/2005/05/00/dn1074/dn1074fig1thm.gif)
![[Figure 2]](http://journals.iucr.org/c/issues/2005/05/00/dn1074/dn1074fig2thm.gif)


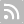
Le développement de la chimie des complexes des métaux de transition à base d'oxalates a pris un essor considérable au cours de la dernière décennie. Ces composés sont intéressants du fait qu'ils peuvent présenter des propriétés physico-chimiques importantes en particulier en magnétisme ou en optique non linéaire (Cheetham et al.,1999). Ils possèdent des charpentes zéolithiques et peuvent présenter une activité catalytique. Ils ont une densité élevée et une bonne stabilité thermique (Kitagowa et al., 1995; Lu et al., 1999). La connaissance des structures cristallines et des mécanismes d'association dans ces complexes, peuvent aider à mieux étudier leurs comportements physiques. Dans ce cadre notre contribution porte sur l'étude de la phase [Cu(C2O4)(C6H16N2)]·4H2O, (I).
La structure est construite à partir du complexe [Cu(C2O4)2(C6H16N2)]. La géométrie de coordination autour de l'atome de cuivre est octaédrique. Dans le plan équatorial sont situés les atomes de ligands O2 et O3 provenant respectivement de deux groupements oxalates et deux atomes d'azote N1 et N2 appartenant à une même molécule organique formant une liaison bidentate. Les deux atomes d'azote N1 et N2 sont en position cis. Ceci est dû à la longueur de la chaîne carbonée. En effet, ils ne sont séparés que par deux atomes de carbone, leur distance est trop courte pour permettre une connexion à l'atome de cuivre en position trans, comme cela se produit dans certains complexes (Jianmin et al.,1995). Les atomes O2, O3, N1 et N2 sont dans un même plan qui contient aussi l'ion Cu2+, sa déviation par rapport à ce plan est seulement de 0.0094 (8) Å. Dans ce plan équatorial, les distances moyennes Cu—O [2.007 (2) Å] et Cu—N [2.062 (2) Å] sont conformes à celles trouvées dans d'autres complexes de cuivre (Cheetham et al., 1999; Jianmin et al., 1995). Les positions axiales sont occupées par deux atomes d'oxygène du groupement oxalate (O1 et O4). La distance moyenne Cu—O axiale est nettement plus longue 2.370 (2) Å e t l'angle O1—Cu—O4 de 156.92 (6)° dévie fortement par rapport à la valeur attendue (180°) pour une disposition trans. Cette déformation pourrait être dûe à l'encombrement stérique due à la base organique.
Les groupements oxalates dans la structure étudiée sont centrosymétriques et plans. Les distances C—C et C═O varient respectivement de 1.466 (6) Å à 1.567 (3) Å e t de 1.244 (3) Å à 1.265 (3) Å. Ces valeurs sont de même ordre de grandeur que celles relevées pour les mêmes groupements, dans des composés analogues (Jianmin et al.,1995; Hokelek et al., 2000; Brown, 1976). Ces ions oxalates servent de ponts entre les atomes de cuivre conduisant á une chaine en zigzag [Cu(C2O4)(C6H16N2)]n progressant le long de la direction b (Fig. 1). Cette forme en zigzag est la conséquence de la disposition des groupements oxalates autour du cation Cu2+. En effet, les plans moyens entre les deux ligands oxalates connectés au cation Cu2+ forment un angle de 76.84 (7)°.
La cohésion de l'édifice cristallin est assurée par des liaisons hydrogène de type O—H···O. Ces liaisons engagent les molécules d'eau et les atomes d'oxygène O1 et O4 des groupements oxalates. Parmi les quatre molécules d'eau, H2OW1 e t H2OW4 sont liées aux groupements oxalates par les ponts hydrogène OW1—H···O4 et OW4—H···O1. Les deux autres molécules d'eau, H2OW2 e t H2OW3, sont liées entre elles et aux molécules H2OW1 e t H2OW4 respectivement. L'ensemble de ces liaisons hydrogènes construit un réseau tridimensionnel complexe.
La comparaison de la structure étudiée, avec celles de complexes de cuivre relevés dans la bibliographie montre que le composé Cu2(C2O4)(C6H16N2)2(OH)2 (Hokelek et al., 2000) renferme les mêmes entités que le composé étudié: cation métallique Cu2+, ion oxalate C2O42− et la base N,N,N',N'-tétraméthyléthylènediamine. Les groupements oxalates dans les deux structures sont centrosymétriques. Les distances interatomiques dans les deux composés sont également comparables. Malgré cette similitude, les deux composés présentent une importante différence, l'une est macromoléculaire (ce travail), l'autre est moléculaire Cu2(C2O4)(C6H16N2)2(OH)2 (Hokelek et al., 2000). Dans ce dernier, le groupement oxalate forme une liaison bidentate avec un seul cation Cu2+ donnant lieu à la formation de dimères. Ceci a vraisemblablement été favorisé par la stoechiométrie des préparations: le rapport C2O4 / Cu étant égal à 2 dans notre cas contre C2O4/Cu = 1 dans le composé de la bibliographie. L'excès du nombre des groupements oxalates par rapport aux atomes de cuivre a favorisé la condensation du complexe dans le composé étudié. La phase Cu2(C2O4)(C6H16N2)2(OH)2 diffère également par l'existence d'une liaison Cu—OH. La formation de cette liaison peut s'expliquer par les précurseurs utilisés. L'acide L-ascorbique permettant la protonation du méthoxyde de cuivre (II).